Atividade 2
2.1 Criando arquivos no sistema eMOF
Nesta atividade, continuaremos utilizando os dados modificados do
dataset iris do pacote vegan com os dados
coletados por Edgar Anderson.
- Anderson E (1935) The irises of the Gaspe Peninsula, Bulletin of the American Iris Society, 59, 2–5.
2.1.1 Checando os dados
O primeiro passo é identificar os campos presentes no dataset:
- Usar a função
strpara conferir os tipos de dados em cada coluna - Aplicar a função
uniquea cada coluna usando a outra função de looplapply
## $Sepal.Length
## [1] 5.1 5.4 5.0 4.7 5.2 4.4 4.5 4.6 7.0 5.5 6.3 6.0 6.7 6.2 6.1 6.4 5.8 5.7 6.5
## [20] 7.3 7.7 7.2 7.9 6.8 5.9 4.8 4.9 6.9 6.6 5.6 7.4 4.3 5.3 7.1 7.6
##
## $Sepal.Width
## [1] 3.5 3.4 3.3 3.2 4.1 3.0 2.3 3.8 2.7 2.2 3.1 2.8 2.9 2.4 2.5 2.6 3.9 4.0 3.6
## [20] 3.7 4.2 2.0 4.4
##
## $Petal.Length
## [1] 1.4 1.7 1.6 1.5 1.2 1.3 1.9 4.7 4.0 3.9 4.4 4.5 4.3 5.0 3.8 5.1 4.2 3.0 5.8
## [20] 6.3 5.5 6.7 6.0 5.6 6.4 5.9 1.0 4.9 4.6 3.6 4.1 4.8 3.5 6.1 5.3 6.9 5.7 5.4
## [39] 5.2 1.1 3.3 3.7 6.6
##
## $Petal.Width
## [1] 0.3 0.2 0.5 0.4 0.1 1.4 1.3 1.6 1.0 1.5 1.7 1.1 1.2 1.9 2.2 1.8 2.0 2.1 2.4
## [20] 2.3 0.6 2.5
##
## $Species
## [1] "Iris setosa" "Iris versicolor" "Iris virginica"
##
## $amostra
## [1] "A18" "A21" "A24" "A27" "A30" "A33" "A36" "A39" "A42" "A45"
## [11] "A48" "A51" "A54" "A57" "A60" "A63" "A66" "A69" "A72" "A75"
## [21] "A78" "A81" "A84" "A87" "A90" "A93" "A96" "A99" "A102" "A105"
## [31] "A108" "A111" "A114" "A117" "A120" "A123" "A126" "A129" "A132" "A135"
## [41] "A138" "A141" "A144" "A147" "A150" "A3" "A6" "A9" "A12" "A15"
## [51] "A17" "A20" "A23" "A26" "A29" "A32" "A35" "A38" "A41" "A44"
## [61] "A47" "A50" "A53" "A56" "A59" "A62" "A65" "A68" "A71" "A74"
## [71] "A77" "A80" "A83" "A86" "A89" "A92" "A95" "A98" "A101" "A104"
## [81] "A107" "A110" "A113" "A116" "A119" "A122" "A125" "A128" "A131" "A134"
## [91] "A137" "A140" "A143" "A146" "A149" "A2" "A5" "A8" "A11" "A14"
## [101] "A19" "A22" "A25" "A28" "A31" "A34" "A37" "A40" "A43" "A46"
## [111] "A49" "A52" "A55" "A58" "A61" "A64" "A67" "A70" "A73" "A76"
## [121] "A79" "A82" "A85" "A88" "A91" "A94" "A97" "A100" "A103" "A106"
## [131] "A109" "A112" "A115" "A118" "A121" "A124" "A127" "A130" "A133" "A136"
## [141] "A139" "A142" "A145" "A148" "A1" "A4" "A7" "A10" "A13" "A16"
##
## $lat
## [1] 48.93597 48.54050 48.32984
##
## $lon
## [1] -64.84098 -64.76651 -65.51233
##
## $date
## [1] "1929-12-01" "1930-02-13"
##
## $site
## [1] "Site1" "Site2" "Site3"Aqui podemos identificar que temos 150 amostras e nomes de espécies associados às amostras, além de atributos (e.g., Petal.Length, Sepal.Width) associados a cada espécie. Também existem dados de coordenadas dos sites e datas das amostragens.
Podemos aproveitar para investigar algumas características dos dados e fazer um controle de qualidade. Por exemplo, verificar se existem valores discrepantes nas variáveis numéricas ou se existem valores faltantes.
Inicialmente, vamos fazer uma inspeção visual da distribuição dos valores numéricos.
iris %>%
select(Species, Sepal.Length:Petal.Width) %>%
pivot_longer(cols = -Species, names_to = "variavel", values_to = "valores") %>%
ggplot(aes(x = valores, fill = Species)) +
geom_histogram() +
facet_wrap(~ variavel, scales = 'free_x') +
theme_classic() +
theme(legend.position = "bottom") +
labs(x = "tamanho (mm)") +
scale_fill_discrete(
expression(bold("Species:")),
labels = c(expression(italic("Iris setosa")),
expression(italic("Iris versicolor")),
expression(italic("Iris virginica"))))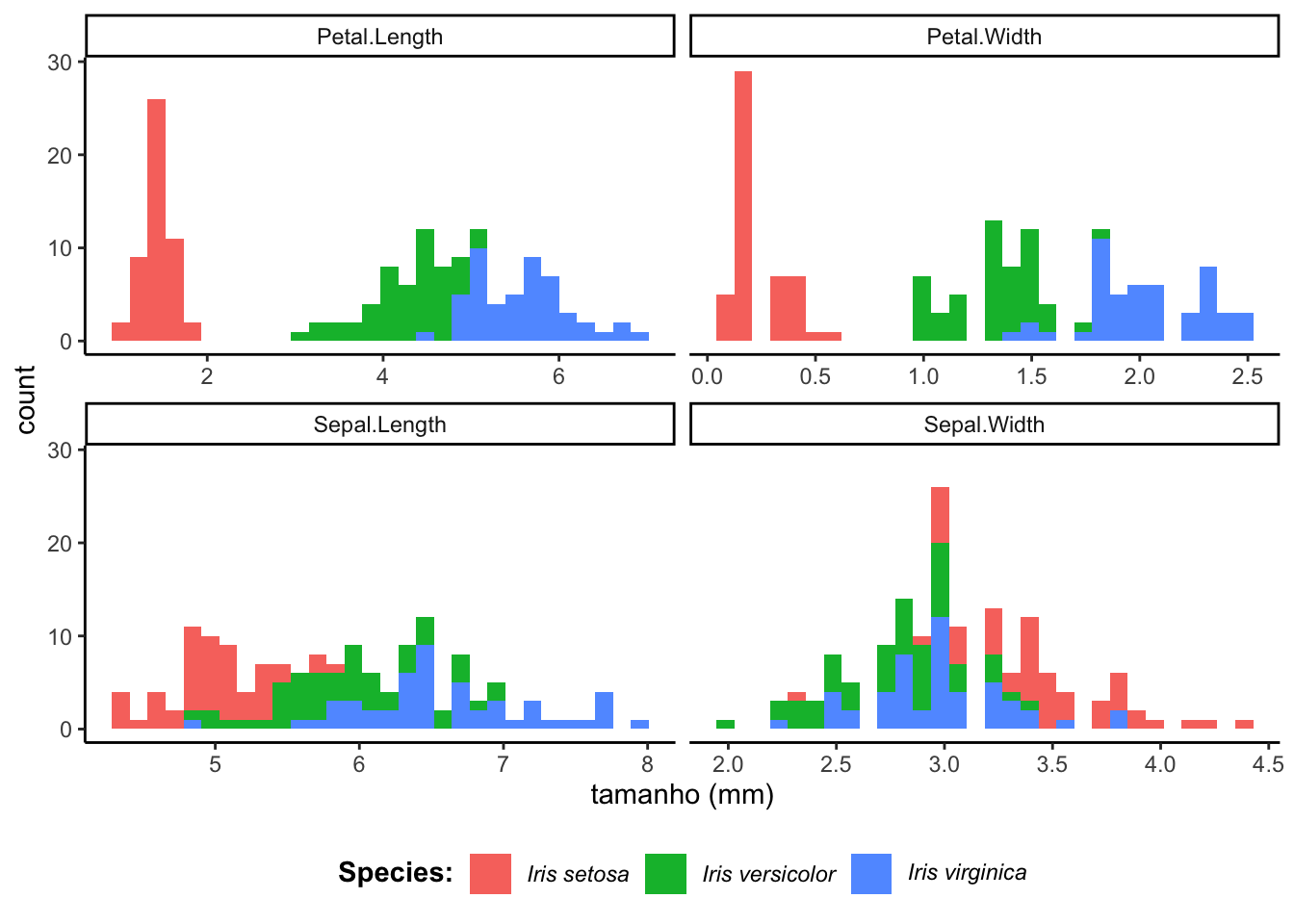
Aqui podemos observar a variação dos dados que tendem a tem distribuição unimodal, mas sem valores extremos.
Para checar as demais variáveis, vamos utilizar o pacote
validate. Neste pacote é possível indicar os padrões
esperados de cada variável e verificar quais observações violam os
limites indicados. Para isso são definidas as regras (e.g. valores de
latitude entre -90 e 90, variável site como
caracter). Para mais informações, basta acessar o vignette
do pacote validate
rules <- validator(in_range(lat, min = -90, max = 90),
in_range(lat, min = -180, max = 180),
is.character(site),
is.numeric(date),
all_complete(iris))
out <- confront(iris, rules)
summary(out)## name items passes fails nNA error warning
## 1 V1 150 150 0 0 FALSE FALSE
## 2 V2 150 150 0 0 FALSE FALSE
## 3 V3 1 1 0 0 FALSE FALSE
## 4 V4 1 0 1 0 FALSE FALSE
## 5 V5 1 1 0 0 FALSE FALSE
## expression
## 1 in_range(lat, min = -90, max = 90)
## 2 in_range(lat, min = -180, max = 180)
## 3 is.character(site)
## 4 is.numeric(date)
## 5 all_complete(iris)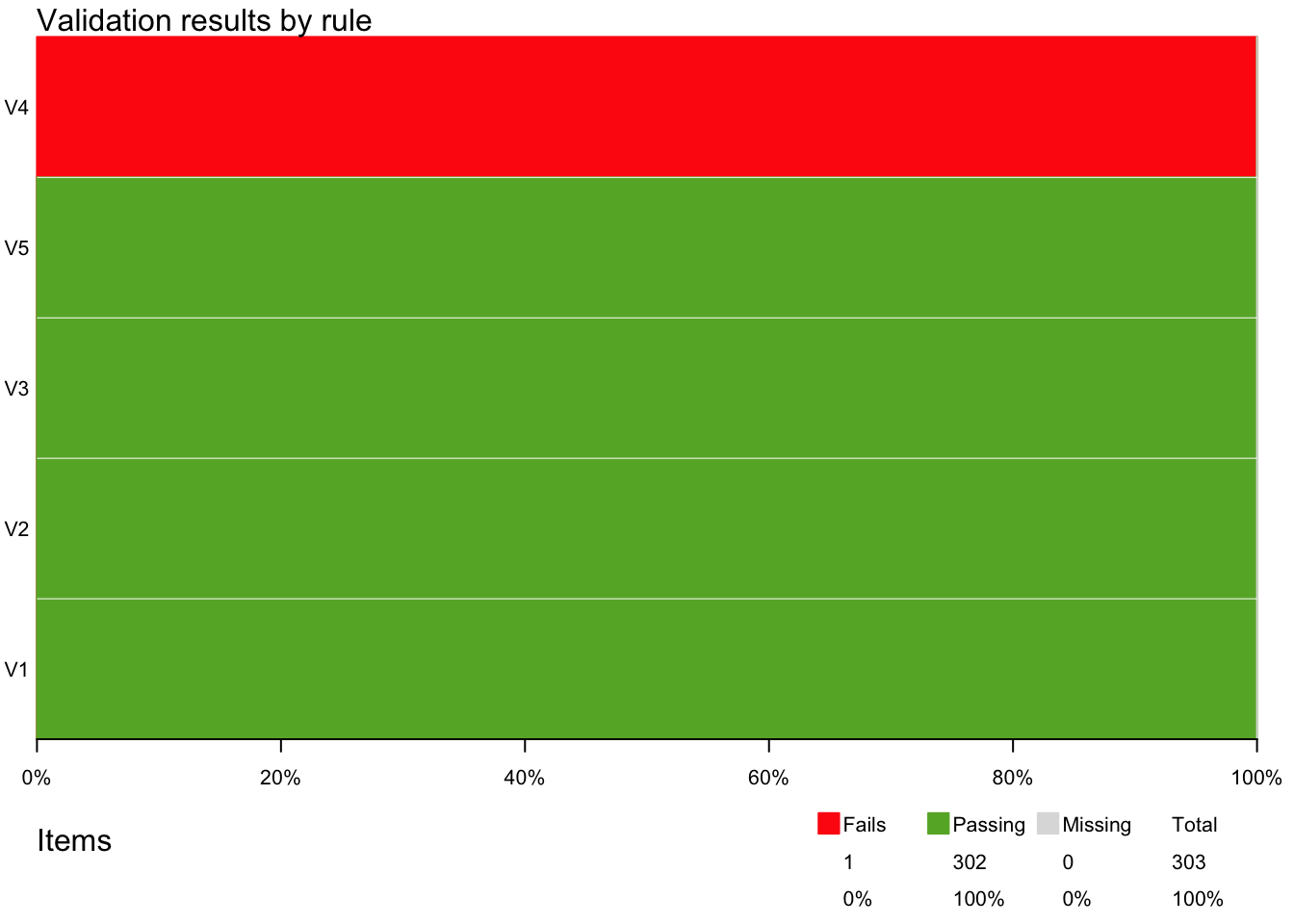
2.1.2 Taxóns
Avaliando os dados, podemos ver que não existem inconsistências e que já possuímos quase todos os campos obrigatórios para o sistema Darwin Core (DwC).
Em seguida, um passo importante é checar se os nomes utilizados dos táxons são válidos.
- Para isso podemos utilizar a função
filter_namedo pacotetaxadb. Esta função permite investigar os nomes em diferentes base de dados (e.g. GBIF, ITIS, COL etc.), mas neste caso vamos buscar na Integrated Taxonomic Information System se a nomenclatura aplicada aos táxons é válida ou se necessita de atualização.
# check taxa
species <- iris %>%
distinct(Species) %>%
pull() %>%
c("Iris murchosa", .) %>% # inserimos uma espécie fictícia para teste
filter_name(., provider = "itis") %>%
data.frame() %>%
bind_cols(Species = iris %>%
distinct(Species) %>%
pull())Podemos observar que nossa impostora (Iris murchosa) não existe na base de dados do ITIS.
Os campos de match e pattern_match indicam
que as espécies são válidas e, o campo uri indica o
endereço único para checar a espécie no ITIS. Este campo
uri será utilizado na planilha occurrence do
sistema event core.
2.1.3 Manipulando os dados
2.1.3.1 Planilha base
Sabendo-se que podemos prosseguir sem corrigir os táxons, agora é necessário renomear as variáveis de acordo com o DwC.
O primeiro passo é criar uma identidade única para as menores unidades de ocorrência dos táxons (
occurrenceID) e das amostras (eventID).Em seguida são adicionadas as informações de espécie (
uri) e renomeados os campos de acordo com o DwC.Por fim, são adicionados campos recomendados que detalham as variáveis presentes no
dataset(eg. sistema de coordenadas, datum, amostrador etc)
OBS: os dados das coordenadas são fictícios e o nome do amostrador é baseado em informação do pacote
vegan(para checar, carregue o pacotevegane digite?iris)
iris_1 <- iris %>%
dplyr::mutate(eventID = paste(site, date, sep = "_"), # create indexing fields
occurrenceID = paste(site, date, amostra, sep = "_")) %>%
left_join(species %>%
select(Species, acceptedNameUsageID, scientificName)) %>% # add species unique identifier
dplyr::rename(decimalLongitude = lon, # rename fields according to DwC
decimalLatitude = lat,
eventDate = date) %>%
mutate(geodeticDatum = "WGS84", # and add complimentary fields
verbatimCoordinateSystem = "decimal degrees",
georeferenceProtocol = "Random coordinates obtained from Google Earth",
locality = "Gaspe Peninsula",
recordedBy = "Edgar Anderson",
taxonRank = "Species",
organismQuantityType = "individuals",
basisOfRecord = "Human observation")2.1.3.2 Planilhas do eMOF
Com os campos adicionados na planilha já podemos iniciar a construção das três matrizes necessárias para inserir os dados em repositórios baseados em ocorrências e dados acessórios como o GBIF.
Os campos obrigatórios na planilha de eventos
(eventCore) são: * eventID = indica os eventos únicos de
amostragem * eventDate = data da amostragem no formato YYYY-MM-DD (ano
com 4 dígitos, mês com 2 dígitos e dia com 2 dígitos) * decimalLongitude
= longitude indicada em graus decimais * decimalLatitude = latitude
indicada em graus decimais * verbatimCoordinateSystem = tipo de
coordenadas * geodeticDatum = Datum das coordenadas
Os termos padronizados do DwC podem ser encontrados no site do DwC.
2.1.3.2.1 Planilha de eventos
Nesta planilha, apenas os campos relacionados às amostras e características gerais do conjunto de dados são selecionadas.
2.1.3.2.2 Planilha de ocorrências
O próximo passo é derivar as ocorrências únicas presentes no dataset. Como ocorrência deve ser considerada a ocorrência única de determinada espécie.
2.1.3.2.3 Planilha de atributos (eMOF)
Por fim, podemos associar atributos a cada ocorrência, neste caso, medidas morfométricas das flores das espécies indicadas. Aqui também devem ser adicionados os campos onde são detalhados os tipos de atributos associados às ocorrências, por exemplo, indicar que as medidas foram tomadas de indivíduos e medidas em centímetros. Os nomes das medidas foram renomeados para seguir padrões de boas práticas.
## create measurementsOrFacts
eMOF <- iris_1 %>%
select(eventID, occurrenceID, recordedBy, Sepal.Length:Petal.Width) %>%
pivot_longer(cols = Sepal.Length:Petal.Width,
names_to = "measurementType",
values_to = "measurementValue") %>%
mutate(measurementUnit = "cm",
measurementType = plyr::mapvalues(measurementType,
from = c("Sepal.Length", "Sepal.Width", "Petal.Width", "Petal.Length"),
to = c("sepal length", "sepal width", "petal width", "petal length")))2.1.3.3 Controle de qualidade
O penúltimo passo é um rápido controle de qualidade para checar se
todas as planilhas tem os mesmos valores de eventID.
Afinal, este campo é o elo entre todas as planilhas.
## character(0)## character(0)## character(0)## # A tibble: 0 × 6
## # ℹ 6 variables: eventID <chr>, occurrenceID <chr>, recordedBy <chr>,
## # measurementType <chr>, measurementValue <dbl>, measurementUnit <chr>## [1] eventID occurrenceID scientificName
## [4] acceptedNameUsageID recordedBy taxonRank
## [7] organismQuantityType basisOfRecord
## <0 rows> (or 0-length row.names)2.1.3.4 Escrevendo as matrizes como arquivos de texto
Tudo parece OK! Agora podemos remover os arquivos intermediários e
deixar apenas os que serão publicados, para então salvá-los como arquivo
*.csv.
rm(list = setdiff(ls(), c("eventCore", "occurrences", "eMOF")))
files <- list(eventCore, occurrences, eMOF)
data_names <- c("DF_eventCore","DF_occ","DF_eMOF")
dir.create("Dwc_Files")
for(i in 1:length(files)) {
path <- paste0(getwd(), "/", "DwC_Files")
write.csv(files[[i]], paste0(path, "/", data_names[i], ".csv"))
}Agora já está tudo pronto para subir os dados para o repositório ou compartilhar diretamente. Só não esqueça dos metadados!!
Colabore, compartilhe, e cite as fontes!